Криобанк культур клеток - CellZoo
 Уникальная научная установка “Криобанк культур клеток” (неофициальное название – “CellZoo”) создана на базе отдела разнообразия и эволюции геномов Института молекулярной и клеточной биологии СО РАН в соответствии с приказом директора ИМКБ №9 от 20 февраля 2024 г.
Уникальная научная установка “Криобанк культур клеток” (неофициальное название – “CellZoo”) создана на базе отдела разнообразия и эволюции геномов Института молекулярной и клеточной биологии СО РАН в соответствии с приказом директора ИМКБ №9 от 20 февраля 2024 г.
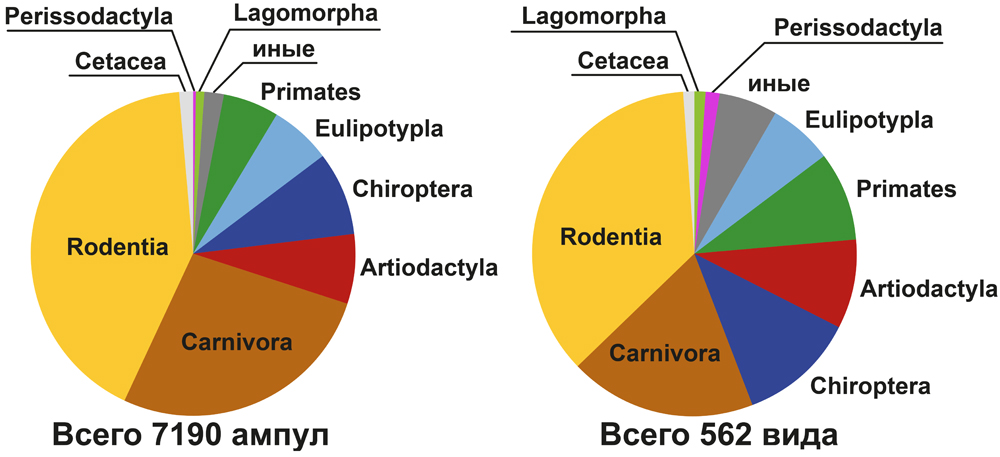
По состоянию на 1 января 2026 года “Криобанк культур клеток” насчитывает 8500 единиц хранения, включающих культуры клеток (8163 ампулы), криоконсервированные ткани, пригодные для получения культур клеток (245 ампул), и биологические материалы, подготовленные для иных исследований (92 единицы).
Культуры клеток получены для 678 видов и 21 гибрида, представляющих классы млекопитающих (562 вида и 20 гибридов), птиц (52 вида), рептилий (42 вида), амфибий (3 вида) и рыб (19 видов и 1 гибрид), которые собраны в основном на территории России и сопредельных стран.
Списки культур
 |
 |
 |
|
Млекопитающие |
Птицы |
Рептилии |
 |
 |
 |
| Амфибии | Рыбы | Ткани |
Культуры клеток млекопитающих
Уникальность коллекции
По видовому разнообразию биоматериалы “Криобанка культур клеток” являются уникальной и одной из крупнейших в мире биоресурсной коллекцией тканей и культур фибробластов и не имеет аналогов подобного масштаба на территории РФ. Большинство видов, материалы которых хранятся в криобанке, отсутствуют в других аналогичных крупнейших коллекциях (Смитсоновский институт, Вашингтон; Научный департамент зоопарка Сан-Диего; Центр клеточных культур Канмин, КНР).
Оборудование УНУ
Для хранения биоматериалов и их поддержания используют стерильные боксы, оборудованные ламинарными шкафами, СО2-инкубаторами, холодильниками, центрифугами и пр.; низкотемпературные холодильники и криохранилища для длительного хранения образцов. С перечнем оборудования можно ознакомиться в разделе “Документы, регламентирующие деятельность УНУ”.
Роль УНУ в стратегии развития генетических технологий
“Криобанк культур клеток” обеспечивает фундамент для широкого ряда биологических и междисциплинарных исследований сразу по нескольким научным направлениям Федеральной научно-технической программы развития генетических технологий на 2019-2027 годы, утвержденной постановлением Правительства Российской Федерации от 22.04.2019 № 479:
• “Биобезопасность и обеспечение технологической независимости”, а именно “создание генетической базы данных биологических объектов, содержащей геномные данные не менее чем 2500 организмов”;
• “Генетические технологии для медицины”;
• “Генетические технологии для развития сельского хозяйства”, а именно “разработка генетических технологий, применяемых в животноводстве и аквакультуре”.
Основные направления исследований, проводимых в УНУ
• Совершенствование стандартов сбора, транспортировки и хранения ценных образцов генетических ресурсов;
• Развитие методик получения и культивирования клеточных культур, а также характеризации биоматериалов;
• Характеризация биоматериалов позвоночных мировой фауны.
Перечень выполняемых типовых работ и оказываемых услуг
• Кариотипический анализ клеточных линий;
• Получение краткосрочных культур лимфоцитов человека;
• Получение первичных культур фибробластов животных;
• Наращивание культур клеток для выделения ДНК и РНК и для других целей;
• Консультация по работе с культурами клеток и получению суспензий хромосом, кариотипическому анализу и молекулярно-цитогенетическим методам.
С перечнем выполняемых работ и оказываемых услуг с указанием порядка расчета их стоимости можно ознакомиться в разделе “Документы, регламентирующие деятельность УНУ”.
Востребованность коллекции
Материалы Криобанка культур клеток востребованы как зарубежными, так и российскими организациями, а также международными консорциумами по геномным проектам.

Сведения о выполненных работах и оказанных услугах
Публикации, выполненные с использованием материалов и/или оборудования УНУ
С 2017 года до 2 мая 2023 года “Криобанк культур клеток” входил в состав ЦКП “Молекулярная и клеточная биология” под названием “Коллекция культур клеток общебиологического назначения”, а до 20 февраля 2024 года функционировал как самостоятельный Центр коллективного пользования, что было отражено при публикации работ.
-
Tomarovsky AA, Khan R, Dudchenko O, Beklemisheva VR, Perelman PL, Totikov AA, Serdyukova NA, Bulyonkova TM, Pobedintseva M, Abramov AV, Weisz D, Yakupova A, Zhuk A, Graphodatsky AS, Powell R, Lieberman Aiden E, Koepfli K-P, Kliver S. Novel chromosome-length genome assemblies of three distinct subspecies of pine marten, sable, and yellow-throated marten (genus Martes, family Mustelidae). J Hered, 2025, doi: 10.1093/jhered/esaf101
-
Biltueva LS, Lemskaya NA, Abramov AV, Platonov VV, Serdyukova NA. Comparative analysis of karyotypes of white-toothed shrews Crocidura lasiura and Crocidura shantungensis (Eulipotyphla, Mammalia). Mamm Biol, 2025, doi: 10.1007/s42991-025-00548-1
-
Proskuryakova AA, Ivanova ES, Galkina S, Beklemisheva VR, Kulak MM, Tishakova KV, Serdyukova NA, Yudkin VA, O’Connor R, Griffin DK, Graphodatsky AS. Toward a comprehensive overview of cytogenomics in the Galliformes: a focus on western capercaillie and hazel grouse karyotypes. (doi: 10.1159/000545036) Cytogenet Genome Res 165(3-5): 206-218, 2025
-
Lisachova L, Lisachov A, Romanenko S, Davletshina G, Altmanová M, Rovatsos M, Kratochvíl L, Giovannotti M, Nazarov R, Okshtein I, Trifonov V. Concerted evolution of genus-specific centromeric satellite DNA in Eremias (Lacertidae, Reptilia). (doi: 10.1159/000543883) Cytogenet Genome Res 165(3-5): 149-161, 2025
-
Romanenko SA, Prokopov DYu, Marchenko SA, Kulak MM, Ilina AV, Serdyukova NA, Galkina SA, Trifonov VA. In situ and in silico localization of major satellite DNAs in the genome of the red-eared slider (Trachemys scripta elegans, Emydidae, Testudines). (doi: 10.1159/000544908) Cytogenet Genome Res 165(3-5): 162-174, 2025
-
Pavlova SV, Biltueva LS, Lemskaya NA, Serdyukova NA. Detailed characterization of karyotypes of white-toothed shrews Suncus etruscus and Crocidura sicula (Eulipotyphla, Mammalia). (doi: 10.1007/s42991-025-00499-7) Mamm Biol 105(5): 589-598, 2025
-
Tomarovsky A, Khan R, Dudchenko O, Totikov A, Serdyukova NA, Weisz D, Vorobieva NV, Bulyonkova T, Abramov AV, Nie W, Wang J, Romanenko SA, Proskuryakova AA, Cherkasov N, Ferguson-Smith MA, Yang F, Balanovskaya E, Gilbert MTP, Graphodatsky AS, Aiden EL, Powell R, Koepfli KP, Perelman PL, Kliver S. Chromosome length genome assembly of the stone marten (Martes foina, Mustelidae): a new view on one of the cornerstones in carnivore cytogenetics. (doi: 10.1093/jhered/esaf001) J Hered 116(4): 548-557, 2025
-
Pavlova SV, Romanenko SA, Matveevsky SN, Kuksin AN, Dvoyashov IA, Kovalskaya YuM, Proskuryakova AA, Serdyukova NA, Petrova TV. Supernumerary chromosomes enhance karyotypic diversification of narrow-headed voles of the subgenus Stenocranius (Rodentia, Mammalia). (doi: 10.1002/jez.b.23273) J Exp Zool B Mol Dev Evol 342(8): 538-556, 2024
-
Беклемишева ВР, Тишакова КВ, Романенко СА, Андреюшкова ДА, Юдкин ВА, Интересова ЕА, Янг Ф, Фергюсон-Смит МА, Графодатский АС, Проскурякова АА. Комплексный цитогенетический анализ кариотипов трех видов уток (шилохвость, кряква и обыкновенный гоголь) и эволюция кариотипов у представителей семейства Anatidae (Anseriformes, Aves). (doi: 10.18699/vjgb-24-84) Вавиловский журнал генетики и селекции 28(7): 759-769, 2024
-
Kononova Y, Adamenko L, Kazachkova E, Solomatina M, Romanenko S, Proskuryakova A, Utkin Y, Gulyaeva M, Spirina A, Kazachinskaia E, Palyanova N, Mishchenko O, Chepurnov A, Shestopalov A. Features of SARS-CoV-2 replication in various types of reptilian and fish cell cultures. (doi: 10.3390/v15122350) Viruses 15(12): 2350, 2023
-
Romanenko SA, Kliver SF, Serdyukova NA, Perelman PL, Trifonov VA, Seluanov A, Gorbunova V, Azpurua J, Pereira JC, Ferguson-Smith MA, Graphodatsky AS. Integration of fluorescence in situ hybridization and chromosome-length genome assemblies revealed synteny map for guinea pig, naked mole-rat, and human. (doi: 10.1038/s41598-023-46595-x) Sci Rep 13: 21055, 2023
-
Lisachov A, Tishakova K, Romanenko S, Lisachova L, Davletshina G, Prokopov D, Kratochvíl L, O'Brien P, Ferguson-Smith M, Borodin P, Trifonov V. Robertsonian fusion triggers recombination suppression on sex chromosomes in Coleonyx geckos. (doi: 10.1038/s41598-023-39937-2) Sci Rep 13: 15502, 2023
-
Proskuryakova AA, Ivanova ES, Makunin AI, Larkin DM, Ferguson-Smith MA, Yang F, Uphyrkina OV, Perelman PL, Graphodatsky AS. Comparative studies of X chromosomes in Cervidae family. (doi.org/10.1038/s41598-023-39088-4) Sci Rep 13: 11992, 2023
-
Kusliy MA, Yurlova AA, Neumestova AI, Vorobieva NV, Gutorova NV, Molodtseva AS, Trifonov VA, Popova KO, Polosmak NV, Molodin VI, Vasiliev SK, Semibratov VP, Iderkhangai T-O, Kovalev AA, Erdenebaatar D, Graphodatsky AS, Tishkin AA. Genetic history of the Altai breed horses: from ancient times to modernity. (doi: 10.3390/genes14081523) Genes 14(8): 1523, 2023
-
Biltueva LS, Vorobieva NV, Lemskya NA, Perelman PL, Trifonov VA, Panov VV, Abramov AV, Kawada S-i, Serdukova NA, Graphodatsky AS. Chromosomal evolution of the Talpinae. (doi: 10.3390/genes14071472) Genes 14(7): 1472, 2023
-
Beklemisheva VR, Lemskaya NA, Prokopov DY, Perelman PL, Romanenko SA, Proskuryakova AA, Serdyukova NA, Utkin YA, Nie W, Ferguson-Smith MA, Fentang Y, Graphodatsky AS. Maps of constitutive-heterochromatin distribution for four martes species (Mustelidae, Carnivora, Mammalia) show the formative role of macrosatellite repeats in interspecific variation of chromosome structure. (doi: 10.3390/genes14020489) Genes 14(2): 489, 2023
-
Proskuryakova AA, Ivanova ES, Perelman PL, Ferguson-Smith MA, Yang F, Okhlopkov IM, Graphodatsky AS. Comparative studies of karyotypes in the Cervidae family. (doi: 10.1159/000527349) Cytogenet Genome Res 162(6): 312-322, 2022
-
Dumas F, Perelman PL, Biltueva L, Roelke M. Retrotransposon mapping in spider monkey genomes of the family Atelidae (Platyrrhini, Primates) shows a high level of LINE-1 amplification. (doi: 10.4081/jbr.2022.10725) J Biol Res 95(2): 10725, 2022
-
Tishakova KV, Prokopov DY, Davletshina GI, Rumyantsev AV, O’Brien PCM, Ferguson-Smith MA, Giovannotti M, Lisachov AP, Trifonov VA. Identification of Iguania ancestral syntenic blocks and putative sex chromosomes in the veiled chameleon (Chamaeleo calyptratus, Chamaeleonidae, Iguania). (doi: 10.3390/ijms232415838) Int J Mol Sci 23(24): 15838, 2022
-
Romanenko SA, Prokopov DY, Proskuryakova AA, Davletshina GI, Tupikin AE, Kasai F, Ferguson-Smith MA, Trifonov VA. The cytogenetic map of the Nile crocodile (Crocodylus niloticus, Crocodylidae, Reptilia) with fluorescence in situ localization of major repetitive DNAs. (doi: 10.3390/ijms232113063) Int J Mol Sci 23(21): 13063, 2022
-
Beklemisheva VR, Belokopytova PS, Fishman VS, Menzorov AG. Derivation of ringed seal (Phoca hispida) induced multipotent stem cells. (doi: 10.1089/cell.2021.0037) Cell Reprogram 23(6): 326-335, 2021
-
Romanenko SA, Smorkatcheva AV, Kovalskaya YM, Prokopov DY, Lemskaya NA, Gladkikh OL, Polikarpov IA, Serdyukova NA, Trifonov VA, Molodtseva AS, O’Brien PCM, Golenishchev FN, Ferguson-Smith MA, Graphodatsky AS. Complex structure of Lasiopodomys mandarinus vinogradovi sex chromosomes, sex determination, and intraspecific autosomal polymorphism. In: Ruiz-Herrera A, Farré-Belmonte M (eds) Mechanisms Driving Karyotype Evolution and Genomic Architecture, MDPI, Switzerland, 2021, pp 193-209 (doi: 10.3390/books978-3-0365-0157-4)
-
Proskuryakova AA, Kulemzina AI, Perelman PL, Yudkin DV, Lemskaya NA, Okhlopkov IM, Kirillin EV, Farré M, Larkin DM, Roelke-Parker ME, O’Brien SJ, Bush M, Graphodatsky AS. Comparative chromosome mapping of musk ox and the X chromosome among some bovidae species. In: Ruiz-Herrera A, Farré-Belmonte M (eds) Mechanisms Driving Karyotype Evolution and Genomic Architecture, MDPI, Switzerland, 2021, pp 211-224 (doi: 10.3390/books978-3-0365-0157-4)
-
Lisachov AP, Tishakova KV, Romanenko SA, Molodtseva AS, Prokopov DYu, Pereira JC, Ferguson-Smith MA, Borodin PM, Trifonov VA. Whole-chromosome fusions in the karyotype evolution of Sceloporus (Iguania, Reptilia) are more frequent in sex chromosomes than autosomes. (doi: 10.1098/rstb.2020.0099) Philos Trans R Soc Lond B Biol Sci 376(1833): 20200099, 2021
-
Romanenko SA, Malikov VG, Mahmoudi A, Golenishchev FN, Lemskaya NA, Pereira JC, Trifonov VA, Serdyukova NA, Ferguson-Smith MA, Aliabadian M, Graphodatsky AS. New data on comparative cytogenetics of the mouse-like hamsters (Calomyscus Thomas, 1905) from Iran and Turkmenistan. (doi: 10.3390/genes12070964) Genes 12(7): 964, 2021
-
Romanenko SA, Lebedev VS, Bannikova AA, Pavlova SV, Serdyukova NA, Feoktistova NYu, Jiapeng Q, Yuehua S, Surov AV, Graphodatsky AS. Karyotypic and molecular evidence supports the endemic Tibetan hamsters as a separate pergent lineage of Cricetinae. (doi: 10.1038/s41598-021-89890-1) Sci Rep 11: 10557, 2021
-
Bishani A, Prokopov DYu, Romanenko SA, Molodtseva AS, Perelman PL, Interesova EA, Beklemisheva VR, Graphodatsky AS, Trifonov VA. Evolution of tandemly arranged repetitive DNAs in three species of Cyprinoidei with different ploidy levels. (doi: 10.1159/000513274) Cytogenet Genome Res 161: 32-42, 2021
-
Evdokimov A., Popov A., Ryabchikova E., Koval O., Romanenko S., Trifonov V., Petruseva I., Lavrik I., Lavrik O. Uncovering molecular mechanisms of regulated cell death in the naked mole rat. (doi: 10.18632/aging.202577) Aging (Albany NY) 13(3): 3239-3253, 2021
-
Romanenko SA, Fedorova YuE, Serdyukova NA, Zaccaroni M, Stanyon R, Graphodatsky AS. Evolutionary rearrangements of X chromosomes in voles (Arvicolinae, Rodentia). (doi: 10.1038/s41598-020-70226-4) Sci Reports 10: 13235, 2020
-
Lisachov AP, Giovannotti M, Pereira JC, Andreyushkova DA, Romanenko SA, Ferguson-Smith MA, Borodin PM, Trifonov VA. Chromosome painting does not support a sex chromosome turnover in Lacerta agilis Linnaeus, 1758. (doi: 10.1159/000506321) Cytogenet Genome Res 160: 134-140, 2020
-
Atlas of mammalian chromosomes (2nd edition) eds. Graphodatsky AS, Perelman PL, O’Brien SJ. Wiley-Blackwell, USA, 2020, 1008 p
-
Romanenko SA, Smorkatcheva AV, Kovalskaya YM, Prokopov DY, Lemskaya NA, Gladkikh OL, Polikarpov IA, Serdyukova NA, Trifonov VA, Molodtseva AS, O’Brien PCM, Golenishchev FN, Ferguson-Smith MA, Graphodatsky AS. Complex structure of Lasiopodomys mandarinus vinogradovi sex chromosomes, sex determination, and intraspecific autosomal polymorphism.(doi: 10.3390/genes11040374) Genes 11(4): 374, 2020
-
Proskuryakova AA, Kulemzina AI, Perelman PL, Yudkin DV, Lemskaya NA, Okhlopkov IM, Kirillin EV, Farré M, Larkin DM, Roelke-Parker ME, O’Brien SJ, Bush M, Graphodatsky AS. Comparative chromosome mapping of musk ox and the X chromosome among some bovidae species. (doi: 10.3390/genes10110857) Genes 10(11): 857, 2019
-
Romanenko SA, Lyapunova EA, Saidov AS, O’Brien PCM, Serdyukova NA, Ferguson-Smith MA, Graphodatsky AS, Bakloushinskaya I. Chromosome translocations as a driver of persification in mole voles Ellobius (Rodentia, Mammalia). (doi: 10.3390/ijms20184466) Int J Mol Sci 20(18): 4466, 2019
-
Lisachov AP, Makunin AI, Giovannotti M, Pereira JC, Druzhkova AS, Barucchi VC, Ferguson-Smith MA, Trifonov VA. Genetic content of the neo-sex chromosomes in Ctenonotus and Norops (Squamata, Dactyloidae) and degeneration of the Y chromosome as revealed by high-throughput sequencing of inpidual chromosomes. (doi: 10.1159/000497091) Cytogenet Genome Res 157(1-2): 115-122, 2019
-
Bakloushinskaya I, Lyapunova EA, Saidov AS, Romanenko SA, O'Brien PCM, Serdyukova NA, Ferguson-Smith MA, Matveevsky S, Bogdanov AS. Rapid chromosomal evolution in enigmatic mammal with XX in both sexes, the Alay mole vole Ellobius alaicus Vorontsov et al., 1969 (Mammalia, Rodentia). (doi: 10.3897/CompCytogen.v13i2.34224) Comp Cytogenet 13(2): 147-177, 2019
-
Lisachov AP, Galkina SA, Saifitdinova AF, Romanenko SA, Andreyushkova DA, Trifonov VA, Borodin PM. Identification of sex chromosomes in Eremias velox (Lacertidae, Reptilia) using lampbrush chromosome analysis. (doi: 10.3897/CompCytogen.v13i2.34116) Comp Cytogenet 13(2): 121-132, 2019
-
Беклемишева ВР, Мензоров АГ. Использование вектора на основе вируса Сендай для эффективной трансдукции фибробластов ластоногих. (doi: 10.18699/VJ18.445) Вавиловский журнал генетики и селекции 22(8): 1020-1025, 2018
-
Romanenko S, Serdyukova N, Perelman P, Trifonov V, Golenishchev F, Bulatova N, Stanyon R, Graphodatsky A. Multiple intrasyntenic rearrangements and rapid speciation in voles. (doi: 10.1038/s41598-018-33300-6) Sci Reports 8: 14980, 2018
-
Makunin AI, Rajičić M, Karamysheva TV, Romanenko SA, Druzhkova AS, Blagojević J, Vujošević M, Rubtsov NB, Graphodatsky AS, Trifonov VA. Low-pass single-chromosome sequencing of human small supernumerary marker chromosomes (sSMCs) and Apodemus B chromosomes. (doi: 10.1007/s00412-018-0662-0) Chromosoma 127(3): 301-311, 2018
-
Evdokimov A, Kutuzov M, Petruseva I, Lukjanchikova N, Kashina E, Kolova E, Zemerova T, Romanenko S, Perelman P, Prokopov D, Seluanov A, Gorbunova V, Graphodatsky A, Trifonov V, Khodyreva S, Lavrik O. Naked mole rat cells display more efficient excision repair than mouse cells. (doi: 10.18632/aging.101482) Aging (Albany NY) 10: 1454-1473, 2018
-
Romanenko SA, Serdyukova NA, Perelman PL, Pavlova SV, Bulatova NS, Golenishchev FN, Stanyon R, Graphodatsky AS. Intrachromosomal rearrangements in rodents from the perspective of comparative region-specific painting. (doi: 10.3390/genes8090215) Genes 8(9): 215, 2017
-
Proskuryakova AA, Kulemzina AI, Perelman PL, Makunin AI, Larkin DM, Farré M, Kukekova AV, Johnson JL, Lemskaya NA, Beklemisheva VR, Roelke-Parker ME, Bellizzi J, Ryder OA, O’Brien SJ, Graphodatsky AS. X chromosome evolution in Cetartiodactyla. (doi: 10.3390/genes8090216) Genes 8(9): 216, 2017
-
Poplavskaya NS, Romanenko SA, Serdyukova NA, Trifonov VA, Yang F, Nie W, Wang J, Bannikova AA, Surov AV, Lebedev VS. Karyotype evolution and phylogenetic relationships of Cricetulus sokolovi Orlov et Malygin 1988 (Cricetidae, Rodentia) inferred from chromosomal painting and molecular data. (doi: 10.1159/000477521) Cytogenet Genome Res 152: 65-72, 2017